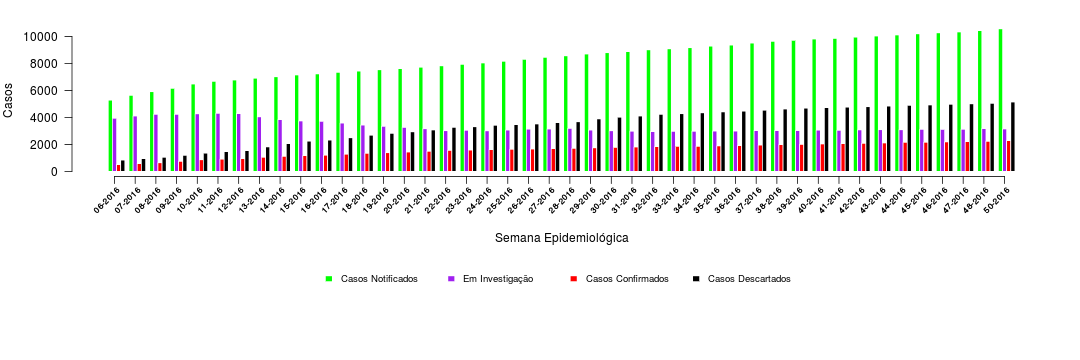
Reproduzindo gráfico de colunas do MS sobre zika/microcefalia (Parte 1, no R)
Já faz alguns dias, durante uma conversa casual em que expunha minha insatisfação em relação às dificuldades para se encontrar dados abertos em formato acessível através de sites de órgãos públicos, fiquei sabendo da existência de alguns portais do governo que, apesar de ainda não atenderem a todas as minhas expectativas, já ajudam bastante. O primeiro é o SAGE (Sala de Apoio à Gestão Estratégica/MS). O segundo é o Portal Brasileiro de Dados Abertos.
Um dado interessante que o SAGE possui é o número de casos notificados, em investigação, confirmados e descartados de microcefalia e/ou alterações do sistema nervoso central (SNC), sugestivos de infecção congênita por zika vírus em fetos, abortamentos, natimortos ou recém-nascidos.
O gráfico original disponível no site é este aqui (clique para ver em tamanho maior).
Para encontrá-lo clique na aba ‘Situação de Saúde’ e depois em ‘Microcefalia e Zika’ no menu que irá aparecer.
Pensei em reproduzir esse gráfico tanto em R como em Python pra que vocês possam aprender um tipo de gráfico que tem algumas peculiaridades e não é tão fácil de fazer, além de poder comparar as duas linguagens ao realizarem a mesma tarefa. Deixo claro aqui que a linguagem que uso há mais tempo é o R (que também é minha favorita! hehe). Comecei a aprender Python este ano depois de muita resistência, então é possível que, de repente, o código do Python ainda possa ser otimizado. Você pode baixar o script em R utilizado para esse post aqui. Para saber qual o tamanho real do código necessário para fazer este gráfico olhe o script.
Antes de mergulhar no post… você precisa saber que o que será tratado aqui contém conteúdo de nível intermediário-avançado em R. Portanto, se você nunca viu um código R antes ou se ainda está nos primeiros passos na linguagem, minha recomendação é que você não leia esse post, caso contrário irá se asssutar! hahaha Nesse caso, considere se inscrever no meu curso de introdução à programação com R. Se ainda assim você estiver curioso, pode ir em frente, mas eu avisei! Se você já tem fluência no R e deseja aprimorar suas habilidades com base graphics, esse post é para você!
Mais pra frente divulgarei uma outra versão deste post feita com o pacote ggplot2 também.
Bom, vamos ao trabalho.
A primeira coisa que devemos fazer é baixar o arquivo contendo os dados. Baixe o arquivo ‘file.csv’ clicando na engrenagem e depois no botão indicado.
Lendo o arquivo
Iniciamos lendo o arquivo utilizando a função read.csv() estipulando o diretório e o nome do arquivo na máquina. O argumento stringsasfactors=FALSE tem o fim de evitar que colunas contendo caracteres sejam convertidas automaticamente em fatores. Se tudo correr bem você terá carregado os dados em formato de dataframe.
# Lendo o arquivo csv (renomeei o arquivo)
zika <- read.csv('/data/sage_ms_zika_casos_2016.csv',
stringsAsFactors = FALSE)
Nessa etapa vale muito usar as funções str(), head(), tail() e is.na() para detectar qualquer problema com os dados e eventuais necessidades de limpeza de dados ou pré-processamento (data cleaning e data wrangling). Não cobrirei aqui o uso dessas funções, mas veja que usando str() podemos ver que o nosso dataframe é composto de 44 observações (linhas) e 5 variáveis (colunas.). Perceba que a primeira coluna é composta de caracteres (strings) e as outras colunas contém o número de casos registrados (notificados, em investigação, confirmados e descartados), ou seja, são valores numéricos inteiros (integers).
# Estrutura dos dados
str(zika)
## 'data.frame': 44 obs. of 5 variables:
## $ Semana : chr "06-2016" "07-2016" "08-2016" "09-2016" ...
## $ Casos.Notificados: int 5280 5639 5909 6158 6480 6671 6776 6906 7015 7150 ...
## $ Em.Investigação : int 3935 4106 4222 4231 4268 4293 4291 4046 3836 3741 ...
## $ Casos.Confirmados: int 508 583 641 745 863 907 944 1046 1113 1168 ...
## $ Casos.Descartados: int 837 950 1046 1182 1349 1471 1541 1814 2066 2241 ...
# Primeiros 6 valores de cada coluna
head(zika)
## Semana Casos.Notificados Em.Investigação Casos.Confirmados
## 1 06-2016 5280 3935 508
## 2 07-2016 5639 4106 583
## 3 08-2016 5909 4222 641
## 4 09-2016 6158 4231 745
## 5 10-2016 6480 4268 863
## 6 11-2016 6671 4293 907
## Casos.Descartados
## 1 837
## 2 950
## 3 1046
## 4 1182
## 5 1349
## 6 1471
De dataframe a matriz e a função barplot()
Uma das coisas que mais causa dor de cabeça quando se tenta fazer um gráfico de barras no R é que, como no nosso caso, estamos trabalhando com dataframes e acabamos tentando aplicar a função barplot() a um objeto deste tipo e obtemos um erro chato como resposta. No entanto, essa função aceita apenas vetores ou matrizes como argumentos. Sendo assim, vamos copiar as colunas com valores numéricos do nosso dataframe para uma matriz.
# Convertendo o dataframe em matriz, excluindo a primeira coluna (caracteres)
zika_matrix <- as.matrix(zika[,-1])
# Primeiros 6 valores de cada coluna da matriz
head(zika_matrix)
## Casos.Notificados Em.Investigação Casos.Confirmados Casos.Descartados
## [1,] 5280 3935 508 837
## [2,] 5639 4106 583 950
## [3,] 5909 4222 641 1046
## [4,] 6158 4231 745 1182
## [5,] 6480 4268 863 1349
## [6,] 6671 4293 907 1471
Ok, mas ainda não terminamos com essa etapa. Se você tentar construir o gráfico com esta matriz, veja o que você vai obter.
barplot(zika_matrix)
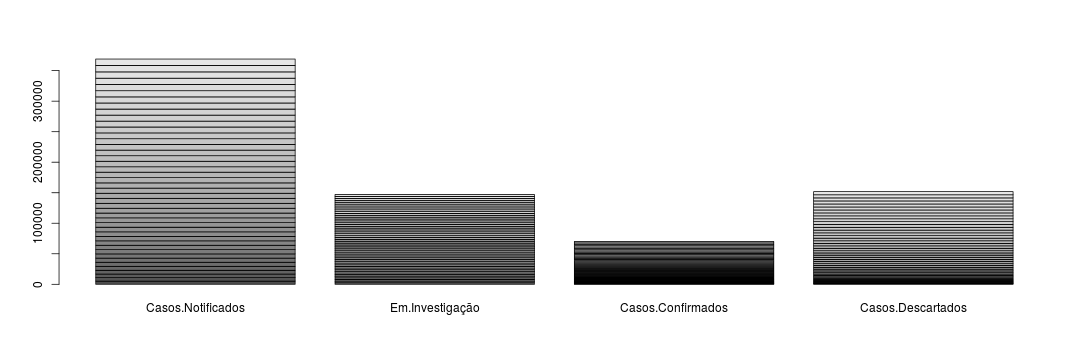
Hmm… não é bem isso que nós desejamos, né. O que aconteceu? O R entendeu que cada valor dentro de uma coluna da matriz era pra ser uma sub-coluna do gráfico. Em outras palavras, ele criou um gráfico de colunas empilhadas. Para obter um gráfico com as colunas lado-a-lado (e não empilhadas) devemos usar o argumento beside=TRUE. Veja a diferença:
barplot(zika_matrix, beside = TRUE)
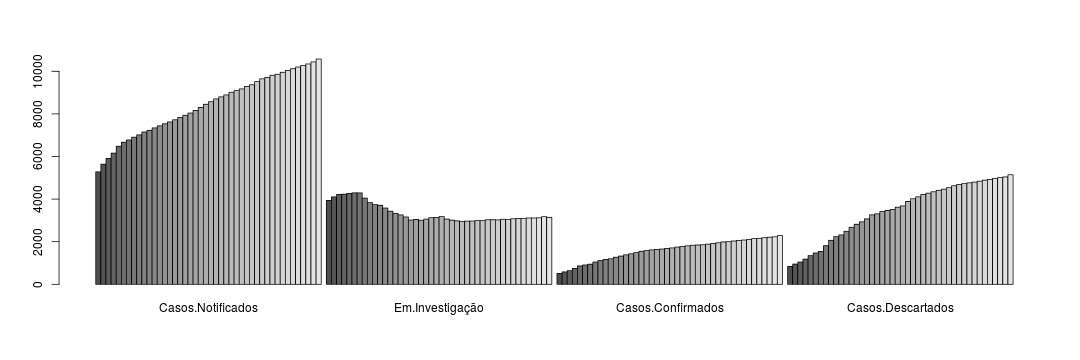
Transpondo a matriz
Mas ainda não é o que queremos. Nós queremos as colunas referentes aos tipos de caso, mas estratificadas pela semana epidemiológica. Para isso, meu amigo, preste atenção, porque a dica que vou passar agora é fundamental e já quebrei muito a cabeça por causa disso. Já que, na hora de gerar o gráfico, o que importa é como as colunas da matriz estão organizadas, nós precisaremos alterar a nossa matriz pra que cada coluna dela seja relativa a uma semana epidemiológica (confira o gráfico original). Para que este conceito fique claro, vou fazer com que os nomes de cada linha da matriz sejam as semanas epidemiológicas, veja:
# Adicionando nomes às linhas da matriz
rownames(zika_matrix) <- zika$Semana
# Primeiras 6 linhas da matriz
head(zika_matrix)
## Casos.Notificados Em.Investigação Casos.Confirmados
## 06-2016 5280 3935 508
## 07-2016 5639 4106 583
## 08-2016 5909 4222 641
## 09-2016 6158 4231 745
## 10-2016 6480 4268 863
## 11-2016 6671 4293 907
## Casos.Descartados
## 06-2016 837
## 07-2016 950
## 08-2016 1046
## 09-2016 1182
## 10-2016 1349
## 11-2016 1471
Continuando nossa explicação dessa parte importante, perceba que na matriz acima as linhas estão relativas às semanas. Mas o que queremos é uma matriz com colunas relativas às semanas. O que fazer? Chamar o chapolim? Não! Basta virar essa matriz de lado! Em outras palavras, deveremos transpor essa matriz usando a função t(). Veja como ela fica agora:
# Transpondo a matriz
zika_matrix <- t(zika_matrix)
# Mostrando as 11 primeiras colunas
# da matriz transposta
zika_matrix[,1:11]
## 06-2016 07-2016 08-2016 09-2016 10-2016 11-2016 12-2016
## Casos.Notificados 5280 5639 5909 6158 6480 6671 6776
## Em.Investigação 3935 4106 4222 4231 4268 4293 4291
## Casos.Confirmados 508 583 641 745 863 907 944
## Casos.Descartados 837 950 1046 1182 1349 1471 1541
## 13-2016 14-2016 15-2016 16-2016
## Casos.Notificados 6906 7015 7150 7228
## Em.Investigação 4046 3836 3741 3710
## Casos.Confirmados 1046 1113 1168 1198
## Casos.Descartados 1814 2066 2241 2320
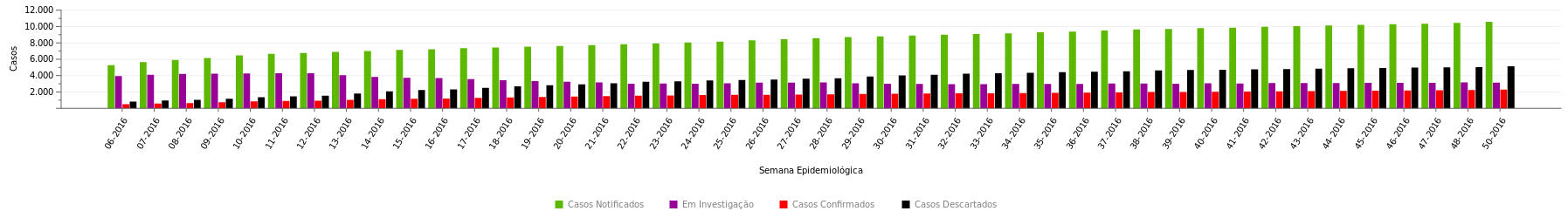
Assim a função barplot vai entender que cada coluna da matriz contém quatro valores, que serão as alturas de quatro colunas lado a lado. Veja como fica o gráfico agora:
# Gráfico de barras
barplot(zika_matrix, beside = TRUE, xaxt = "n")
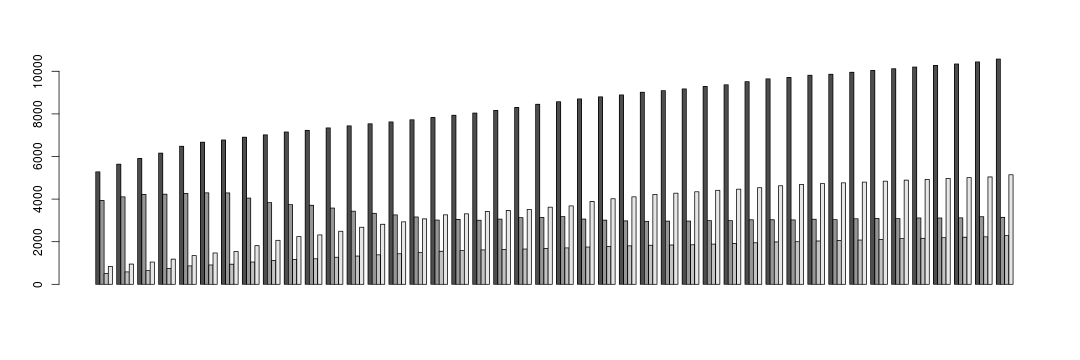
Se você for um leitor atento, verá que adicionei o argumento xaxt="n". Isso quer dizer, ‘tipo do eixo X é igual a nenhum’, ou seja, eu suprimi o eixo X. Fiz Isso porque antes eu havia nomeado as linhas da matriz (as linhas se tornaram colunas na matriz transposta) com as semanas epidemiológicas. Se eu deixasse do jeito que estava o R iria produzir um eixo X automaticamente usando essas semanas e eu não quero isso. Siga em frente que você verá eu adicionando um novo eixo X e aí você entenderá o porquê.
Nosso gráfico está ficando melhor, não? Que tal adicionar um pouco de cor para deixá-lo mais parecido com o original?
# Gráfico de barras
barplot(zika_matrix,
beside = TRUE,
xaxt = "n",
col = c('green', 'purple', 'red', 'black'))
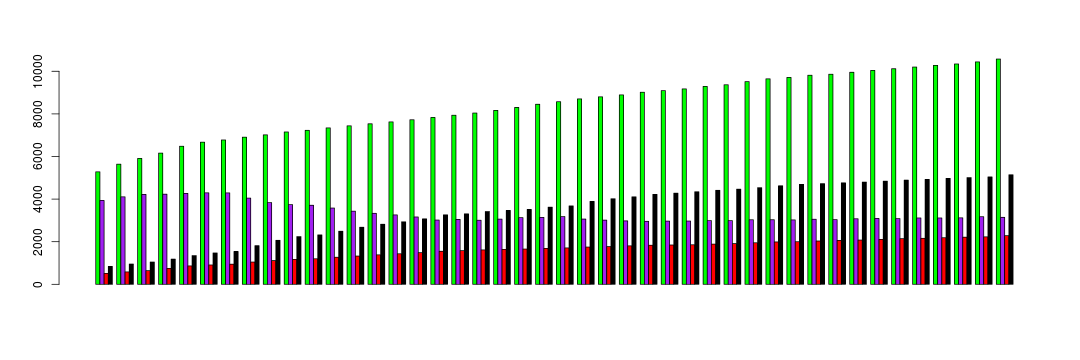
Utilizando o argumento las=1 na função barplot(), podemos alterar a orientação dos números nos eixos:
# Gráfico de barras
barplot(zika_matrix,
beside = TRUE,
xaxt = "n",
col = c('green', 'purple', 'red', 'black'),
las = 1)
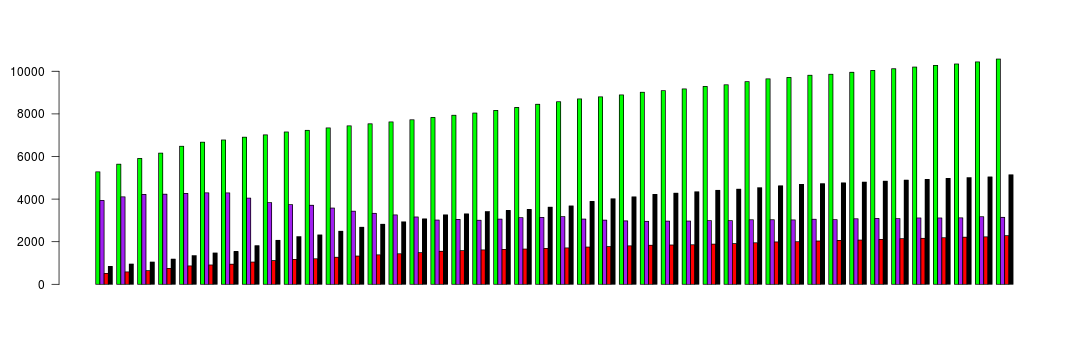
Eixos
Agora nos resta adicionar o eixo X e os títulos dos eixos. Para essa primeira tarefa, usaremos a função axis() e a função text(). Aqui side se refere ao lado em que o eixo deve ser desenhado (1 embaixo, 2 à esquerda, 3 acima e 4 à direita), at às posições das tick marks na linha do eixo, labels é o que deve ser escrito em cada tick mark e line é a posição do eixo (em número de linhas) em relação à margem. O argumento srt=45 utilizado na função text() permite que os textos sejam posicionados com uma angulação de 45 graus.
Perceba que aqui eu também designei o gráfico como uma variável (barp). Por que fiz isso? A função barplot(), além de gerar o gráfico, provê as posições das colunas no eixo X quando designada a uma variável. Por isso, barp agora é um vetor contendo as posições das colunas do gráfico no eixo X. Isso me permite usar essas informações para posicionar corretamente as tick marks e seus textos correspondentes.
Mas antes vamos reservar um espaço extra abaixo para que o eixo X, seu título e tick marks, bem como as legendas (que colocaremos adiante), possam aparecer. Usaremos para este fim a função par() e o seu argumento mar.
# Preparando margens do ambiente gráfico
par(mar = c(13, 5, 2, 2))
# Gráfico de barras
barp <- barplot(zika_matrix,
beside = TRUE,
xaxt = "n",
col = c('green', 'purple', 'red', 'black'),
las = 1)
# Eixo X
axis(side = 1,
at = barp[2,],
labels = rep("",44),
line = .25)
text(x = barp[2,]-2, y = par('usr')[3]-2000,
labels = zika$Semana,
srt=45, xpd = TRUE, cex = .7)
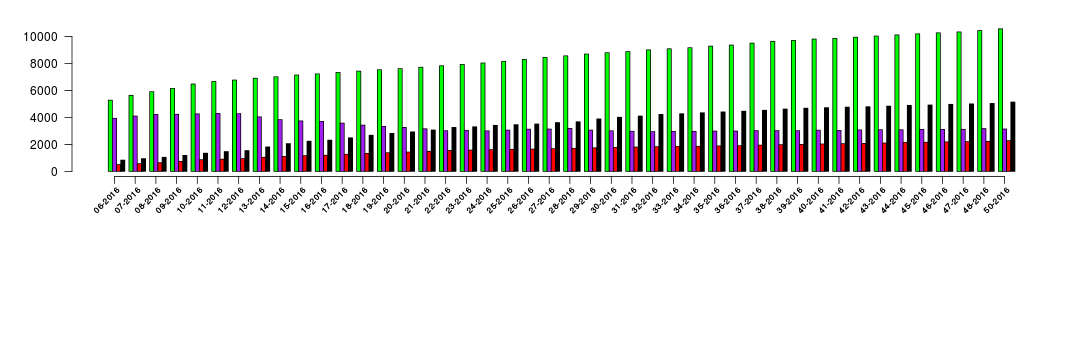
Para nomear os eixos utilizamos as funções title() e os argumentos xlab e ylab. O argumento line aqui tem a mesma função de posicionamento relativo que a utilizada anteriormente.
# Títulos dos eixos X e Y
title(xlab = 'Semana Epidemiológica', line = 4)
title(ylab = 'Casos', line = 4)
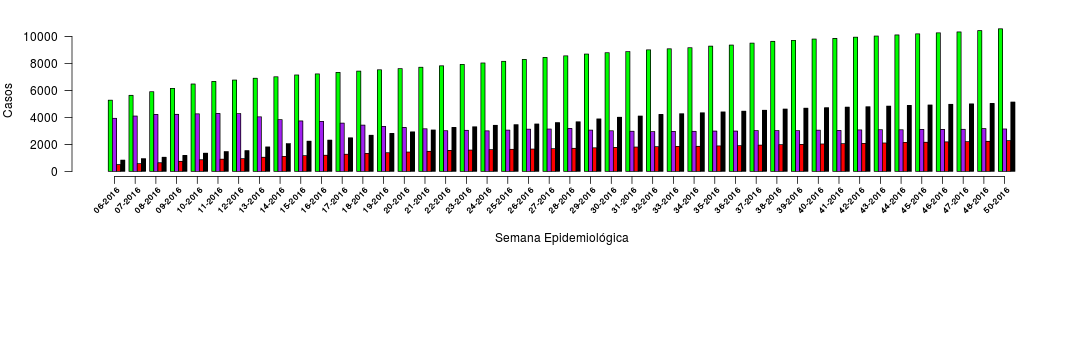
Legendas
Por fim, nos resta adicionar a legenda. Faremos uma legenda de cada vez (ao todo quatro) porque o posicionamento será feito manualmente. Se for feito automaticamente pelo R, não sairá muito legal, vai por mim. Os truques de posicionamento utilizando a função par() e o argumento usr são um tópico bem mais avançado que não cobrirei aqui porque ocuparia muito tempo e espaço. Por enquanto você pode se contentar sabendo que, usando a função legend(), você pode adicionar legendas e que, para atingirmos o máximo de semelhança com o gráfico original da SAGE, precisaremos utilizar os argumentos:
xpara a posição da legenda no eixo X;ypara a posição da lengeda no eixo Y;fillpara definir a cor do quadrado;xpd=TRUEpara permitir a legenda ser plotada fora dos limites do gráfico;horiz=TRUEpara obtermos uma legenda horizontal;bty="n"para eliminarmos a ‘caixa’ que geralmente envolve legendas (bty é uma sigla para box type);cex=.8para definirmos um tamanho de 80% do original para a fonte da legenda.
# Diferença entre coordenadas máxima e mínima do eixo X
diff <- par('usr')[2]-par('usr')[1]
# Legendas
legend(x = par('usr')[1]+diff/4, y = par('usr')[3]-7000,
legend = 'Casos Notificados',
fill = 'green',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8)
legend(x = par('usr')[1]+diff/4+diff/8, y = par('usr')[3]-7000,
legend = 'Em Investigação',
fill = 'purple',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8)
legend(x = par('usr')[1]+diff/4+(diff/8)*2, y = par('usr')[3]-7000,
legend = 'Casos Confirmados',
fill = 'red',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8)
legend(x = par('usr')[1]+diff/4+(diff/8)*3, y = par('usr')[3]-7000,
legend = 'Casos Descartados',
fill = 'black',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8)
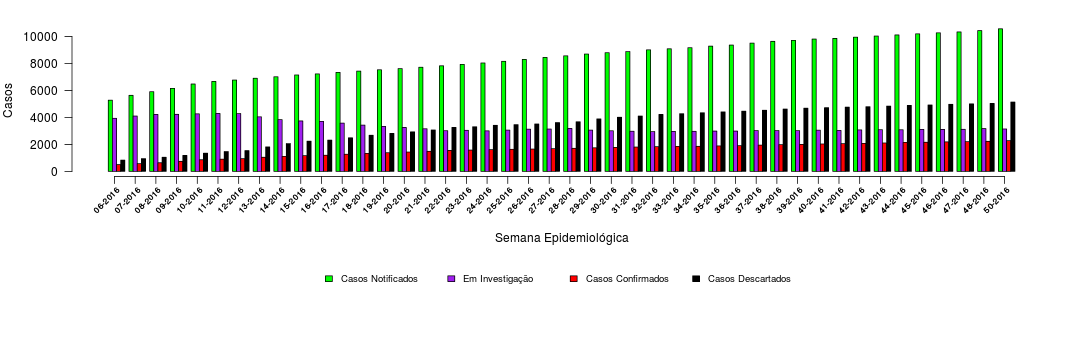
Conclusão
Que tal? Uff… que trabalheira hein… Pois é. Como disse acima, fazer um gráfico desses não é o trabalho mais trivial do mundo. Mas não deixa de ser muito legal! hehehe
Se você gostou desse post e tem interesse em aprender mais sobre R fique de olho no site Programmed Biology para fazer sua inscrição em algum dos meus cursos. Fique à vontade para me enviar uma mensagem particular também no e-mail pmdbio{at}gmail.com.
Tentarei liberar a segunda parte dessa série com o mesmo gráfico reproduzido em Python até Quinta-Feira dessa semana. Não deixe de checar!
Segredo: se você chegou até o final e for muito atento mesmo você perceberá que o gráfico original não possui linhas envolvendo as colunas e nem envolvendo os quadrados das legendas. Para suprimir essas linhas envoltórias, utilizamos o argumento border=FALSE nas funções barplot() e legend():
# Preparando margens do ambiente gráfico
par(mar = c(13, 5, 2, 2))
# Gráfico de barras
barp <- barplot(zika_matrix,
beside = TRUE,
xaxt = "n",
col = c('green', 'purple', 'red', 'black'),
las = 1,
border = FALSE)
# Eixo X
axis(side = 1,
at = barp[2,],
labels = rep("",44),
line = .25)
text(x = barp[2,]-2, y = par('usr')[3]-2000,
labels = zika$Semana,
srt=45, xpd = TRUE, cex = .7)
# Títulos dos eixos X e Y
title(xlab = 'Semana Epidemiológica', line = 4)
title(ylab = 'Casos', line = 4)
# Diferença entre coordenadas máxima e mínima do eixo X
diff <- par('usr')[2]-par('usr')[1]
# Legendas
legend(x = par('usr')[1]+diff/4, y = par('usr')[3]-7000,
legend = 'Casos Notificados',
fill = 'green',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8,
border = FALSE)
legend(x = par('usr')[1]+diff/4+diff/8, y = par('usr')[3]-7000,
legend = 'Em Investigação',
fill = 'purple',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8,
border = FALSE)
legend(x = par('usr')[1]+diff/4+(diff/8)*2, y = par('usr')[3]-7000,
legend = 'Casos Confirmados',
fill = 'red',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8,
border = FALSE)
legend(x = par('usr')[1]+diff/4+(diff/8)*3, y = par('usr')[3]-7000,
legend = 'Casos Descartados',
fill = 'black',
xpd = TRUE, horiz = TRUE, bty = "n", cex = .8,
border = FALSE)